Proteine unter der atomaren Lupe
Gesichter wiedererkennen, zur Straßenbahn laufen oder Haare kämmen – unsere alltäglichen Abläufe sind davon abhängig, dass die Proteine in unserem Körper richtig funktionieren. Verändern sie sich und funktionieren nicht mehr nach Plan, kommt es zu pathologischen Erscheinungen – der Mensch wird krank. Ein interdisziplinäres Projektteam aus Chemiker*innen und Strukturbiolog*innen der Universität Wien und der MFPL nimmt diese Veränderungen nun ganz genau unter die "atomare Lupe": in einem vom WWTF finanzierten Projekt mit dem bezeichnenden Titel "Zooming in on protein functional sites with atomic resolution".
Proteine besser sichtbar machen
Im Projekt werden Methoden entwickelt, die eine Strukturanalyse mit atomarer Auflösung in bestimmten Bereichen ermöglichen. Damit tritt das große Ganze in den Hintergrund und der Blick für das Detail wird geschärft. "Das funktioniert nur in Zusammenarbeit mit der Strukturbiologie, die die Proteine für uns Chemiker sichtbar machen kann", erläutert Projektleiter Christian Becker. Aus diesem Grund haben sich der Chemiker und seine Kolleg*innen Anne Conibear und Roman Lichtenecker mit den beiden Strukturbiolog*innen Kristina Djinovic Carugo und Robert Konrat zusammengetan. Sie möchten Techniken etablieren, die über die Proteinchemie hinaus genutzt werden können.
Modifizierte Proteine
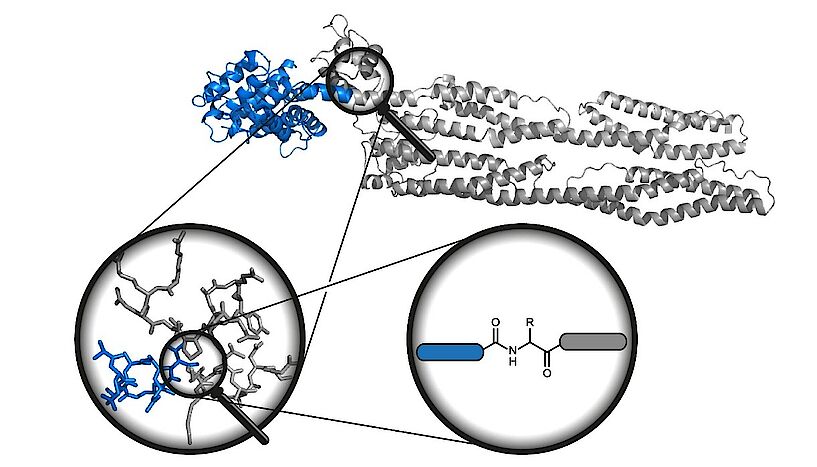
Doch zunächst gilt es, das Ausgangsmaterial herzustellen. Die Gruppe rund um Becker beschäftigt sich schon seit Jahren mit der Herstellung modifizierter Eiweißketten. Für das aktuelle Projekt werden kleine Teile solcher Ketten, sogenannte Peptide, synthetisiert, d.h. künstlich hergestellt. Diese lassen sich recht leicht chemisch modifizieren. Die veränderten Peptide werden in eine größere Proteinstruktur integriert und schließlich mit Methoden der Strukturbiologie analysiert. Dadurch erhofft sich das Projektteam Erkenntnisse über die Auswirkung der Modifikationen.
Das wirklich Besondere liegt aber im Kleinsten: "Ein Protein besteht typischerweise aus hunderten Aminosäuren. Ihre dreidimensionale Anordnung zu ermitteln, ist eine große Herausforderung", erklärt Becker. "Um wirklich nur den Bereich zu treffen, in dem sich Proteine abhängig von Modifikationen verändern, zoomen wir in die Struktur hinein und schauen uns nur einen bestimmten Bereich der Aminosäuren an." Wissen die ForscherInnen mehr über die Strukturveränderung in einem bestimmten Bereich, können sie Rückschlüsse darauf ziehen, wie sich ein modifiziertes Protein im zellulären Kontext verhält.
Chemie und Biologie
Sichtbar werden diese Bereiche durch Methoden aus der Strukturbiologie wie der Kleinwinkelstreuung und der Kernspinresonanz. Mit ihnen können dynamische Prozesse auf Proteinebene bis ins kleinste Detail aufgelöst werden. Damit die relevanten Proteine danach auch wiedergefunden und analysiert werden können, müssen sie zuvor mit einer Isotopen-Markierung versehen werden.
"Mit dieser Methode können wir biologisch relevante Veränderungen in einem Teil der Proteinstruktur nachvollziehen und ihre Auswirkungen auf die Funktionsweise des Proteins analysieren", erklärt Projektpartner Robert Konrat. Methodisch entwickelt wird am Beispiel des Tau-Proteins, das mit Alzheimer in Verbindung steht, und am α-Actinin Protein, das eine wichtige Rolle beim Muskelaufbau spielt.
Neue Wege für Grundlagenforschung
Auch Daten aus früheren Forschungsprojekten helfen den WissenschafterInnen, geeignete Bereiche der Proteine zu selektieren. "Obwohl die Trefferwahrscheinlichkeit bei den ausgewählten Bereichen sehr hoch ist, kann es passieren, dass wir danebenliegen und nachjustieren müssen", erklärt der Projektleiter.
Am Ende dieses Weges soll eine neue Methode stehen, die ForscherInnen auf nahezu jedes Protein anwenden können. Daneben sei das Projekt auch für die angewandte Forschung von Interesse: "Wenn wir lernen, wie sich diese Proteine verändern und warum, können wir die Entstehung von Krankheiten verstehen."
Das Projekt "Zooming in on protein functional sites with atomic resolution – an integrated chemistry approach for structural biology" unter der Leitung von Univ.-Prof. Dr. Christian Friedrich Wilhelm Becker in Zusammenarbeit mit Dr. Anne Conibear und Dr. Roman Lichtenecker von der Fakultät für Chemie sowie mit Univ.-Prof. Dr. Robert Konrat, Group Leader für Computational Biology and Biomolecular NMR Spectroscopy und Univ.-Prof. Dr. Kristina Djinovic Carugo, Group Leader für Structural Biology of the Cytoskeleton an den MFPL, ein Joint Venture der Universität Wien und der Medizinischen Universität Wien, läuft von 1. März 2018 bis 28. Februar 2021 und wird vom WWTF gefördert.
- Institut für Biologische Chemie der Fakultät für Chemie
- Projektwebsite "Structure Zoom"
- WWTF-Projektwebsite
- "Native chemical ligation in protein synthesis and semi-synthesis" in Chemical Society Reviews
- "Single Posttranslational Modifications in the Central Repeat Domains of Tau4 Impact its Aggregation and Tubulin Binding" in Angewandte Chemie